< 필요한 라이브러리 불러오기 >
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
sns.set_theme(style = 'white', context = 'notebook', palette = 'deep')
from collections import Counter
from IPython.core.interactiveshell import InteractiveShell
InteractiveShell.ast_node_interactivity = "all"# sns.set :: 그래프 전체적인 스타일을 조절할 수 있다.
< 데이터 불러오기 >
train = pd.read_csv('C:/Users/82108/Desktop/kaggle/train.csv')
test = pd.read_csv('C:/Users/82108/Desktop/kaggle/test.csv')
IDtest = test['PassengerId']
train.head()
< 이상치 검정 >
# 이상치는 극단적인 영향력을 가지고 있기 때문에 (특히 회귀문제에서), 분석 전에 이상치를 탐색하는 과정이 필요하가.
# 여기서는 Turkey method를 사용하였다 >> boxplot 에서 이상치 나오는 과정을 함수로 만들기
def detect_outliers(df,n,features):
outlier_indices = []
for col in features:
Q1 = np.percentile(df[col],25)
Q3 = np.percentile(df[col],75)
IQR = Q3 - Q1
outlier_step = 1.5*IQR
outlier_list_col = df[(df[col] < Q1 - outlier_step) | (df[col] > Q3 + outlier_step)].index
outlier_indices.extend(outlier_list_col)
outlier_indices = Counter(outlier_indices)
multiple_outliers = list( k for k, v in outlier_indices.items() if v > n)
return multiple_outliers
Outliers_to_drop = detect_outliers(train,2,["Age",'SibSp','Parch','Fare'])# 이상치 검출해주는 함수 detect_outliers()
# 함수 전체적인 설명 :: 각 컬럼별(수치형) 이상치를 검정해서 2개 이상의 컬럼에서 이상치가 있는 관측값의 index를 outlier_indices에 담을 것이다.
# for문을 통해 각 컬럼별 Q1,Q3을 구해 outlier_step을 구한다.
# 각 컬럼별로 이상치 기준점 이하 혹은 이상인 관측값의 index를 outlier_list_col에 저장한다.
# 각 컬렴별로 구한 index를 extend 함수를 통해 합친다.
** append()와 extent()의 차이점은 append는 마지막에 값을 하나씩 붙이는 것이고,
extend를 두 개 이상을 한번에 다 붙 일 수 있다.
# Counter 함수 :: 요소의 개수를 알려주는 함수
# outlier_indices 에는 요소들이 인덱스들로 구성되어 있으니 Counter 함수를 통해 각각의 인덱스가 몇개 있는지 알려준다.
train.loc[Outliers_to_drop]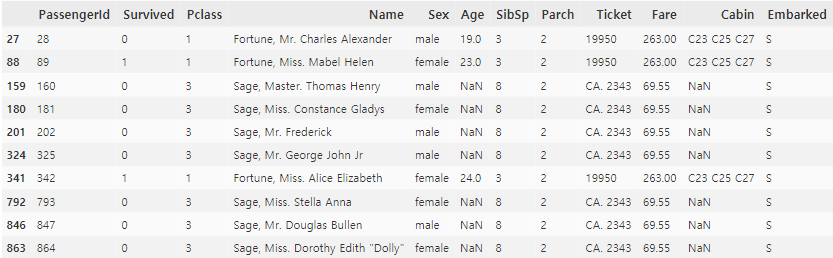
>> 28,89,342 탑승객은 Fare 값이 너무 높고, 나머지 7명은 SisSp가 너무 높다.
train = train.drop(Outliers_to_drop, axis = 0).reset_index(drop = True)# drop() :: row ( axis = 0 )나 columns ( axis = 1 ) 삭제하기
# dropna() :: 결측치가 있는 행을 모두 삭제할 수 있는 함수
# drop_duplicates() :: 중복인 행을 삭제할 수 있는 함수
# reset_index :: 데이터프레임을 먼저 불러온 후, 특정 열을 인덱스로 설정할 때 사용하는 함수
:: drop = True 인덱스로 지정은 하되, 기존열을 남겨놓는다.
>> 이상치로 추출된 관측값들을 제거하자
< train 데이터와 test 데이터 합치기 >
train_len = len(train)
train_len
dataset = pd.concat(objs = [train,test], axis = 0).reset_index(drop = True)
dataset.head()# Feature Engineering을 같이 해주기 위해 train data와 test data 합친다.
# Categorical conversion 하는 과정에서 같은 수의 features을 얻기 위해서
# 예를 들어, train에는 Age변수에 10대 / 20대 / 30대 의 값이 있고, test에는 10~40대 까지 있다면
train으로만 Categorical conversion을 했다면 test에 똑같이 적용하는 과정에서 오류가 발생할 것이다.
< 결측치 확인하기 >
dataset = dataset.fillna(np.nan)
dataset.isnull().sum() 
# train / test 합친 데이터 dataset 의 결측치
# 각 컬럼별 분석 시 채울거임
< Feature analysis >
1. Numerical value
g = sns.heatmap(train[['Survived','SibSp','Parch','Age','Fare']].corr(), annot = True , fmt = ".2f", cmap = 'coolwarm')
>> Fare가 Survived와 상관계수가 가장 높으며 유의해보인다. (0.26)
>> 그렇다고 해서 다른 변수들이 쓸모없다는 것은 절대 아니다
1-1. SibSp vs Survived
g = sns.factorplot(x = 'SibSp', y='Survived' , data = train, kind = 'bar',
size = 6 , palette = 'muted')
g.despine(left = True)
g = g.set_ylabels("survival probability")
# despine() :: 그래프를 좀 더 깔끔하게 그려주도록 도와주는 함수
:: left = True 해줌으로써 왼쪽 축이 없어짐
>> SibSp가 1~2명일 때, 생존율이 높은 편이며 그 이상으로 갈수록 생존율이 떨어진다.
1-2. Parch vs Survived
g = sns.factorplot(x = "Parch", y = 'Survived', data = train , kind = 'bar', size = 6, palette = 'muted')
g.despine(left = True)
g = g.set_ylabels("survival probability")
>> SibSp와 비슷한 패턴을 보임
>> Parch가 1~3명에서 생존율이 높은 편이며, 4 이상의 생존율은 거의 없다.
>> 이를 통해 챙겨야 하는 가족의 수가 많아질수록 생존율이 떨어짐을 짐작할 수 있다.
1-3. Age vs Survived
g = sns.FacetGrid(train, col = 'Survived')
g = g.map(sns.distplot, "Age")
##########################################################
g = sns.kdeplot(train["Age"][(train["Survived"] == 0) & (train["Age"].notnull())], color = 'Red', shade = True)
g = sns.kdeplot(train["Age"][(train["Survived"] == 1) & (train["Age"].notnull())], ax = g , color = 'Blue', shade = True)
g.set_xlabel("Age")
g.set_ylabel("Frequency")
g = g.legend(["Not Survived","Survived"])

# FacetGrid() :: 다중 플롯 그리드를 만들어서 여러가지 쌍 관계를 표현하기 위한 그리드 Class이다. 즉, 도화지에 축을 나누는 것과 같다.
:: FacetGrid(data, row, col, hue) // map함수를 통해 어떤 그래프 그릴것인지 변수명과 함께 명시
# kdeplot() :: 데이터의 분포를 확인하는 커널 밀도 추청 플롯
:: 히스토그램은 구간을 어떻게 설정하느냐에 따라 결과물이 매우 달라진다 >> 대안 >> 커널밀도함수
>> Age 분포는 마치 정규분포와 비슷한 꼴로 나오며, 생존자와 비생존자의 분포가 비슷해보인다.
>> 하지만 큰 차이점은 어린층의 생존율이 훨씬 높음을 알 수 있다.
>> 반대로 60~80세의 생존율은 낮은 편이다.
>> KDE를 보면 어린층에서 그래프가 솟아오른걸 볼 수 있다.
1-4. Fare vs Survived
dataset["Fare"].isnull().sum() # 1개
dataset['Fare'] = dataset["Fare"].fillna(dataset["Fare"].median)>> 결측치가 1개밖에 없으니 Fare의 중간값으로 채움 >> 큰 영향 없을 것이다.
g = sns.distplot(dataset["Fare"], color="m", label="Skewness: %.2f"%(dataset["Fare"].skew()))
g = g.legend(loc="best")에러 나는 거 해결중......
'캐글 필사' 카테고리의 다른 글
| [캐글 필사(1)-1] 타이타닉 EDA & Feature Engineering (0) | 2020.10.11 |
|---|

댓글